Covid-19 : le tableau de bord des variants en France – Le Monde
DécryptagesVisualisez chaque semaine la part des variants du SARS-CoV-2 circulant en France, ainsi que la liste des variants « préoccupants » dressée par l’OMS.
Il y a un an, au Royaume-Uni, la rentrée avait un goût de retour à une quasi-normalité : restaurants et écoles ouvertes, incitations à revenir au bureau… Mais très vite un variant du SARS-CoV-2 va changer la donne. A la fin de septembre, dans le sud-est de l’Angleterre, les laboratoires détectent pour la première fois des mutations génétiques significatives de la souche historique du virus. Aussitôt, Boris Johnson annonce la fermeture à 22 heures des pubs, des cafés et des restaurants anglais. Les autorités britanniques signalent, le 14 décembre, le variant qu’un groupe de chercheurs a identifié et qui présente de nombreuses mutations.
La communauté scientifique sait désormais que le SARS-CoV-2 peut muter. C’était un « phénomène attendu », rappelle Santé publique France, mais le génome de ce coronavirus paraissait jusqu’alors relativement stable. Les chercheurs décèlent par la suite d’autres variants en Afrique du Sud, au Brésil… Depuis la fin de mai 2021, l’Organisation mondiale de la santé (OMS) a choisi de les désigner par une lettre de l’alphabet grec, dénomination jugée moins « stigmatisante » que leur origine géographique.
Quasi-hégémonie du variant Delta
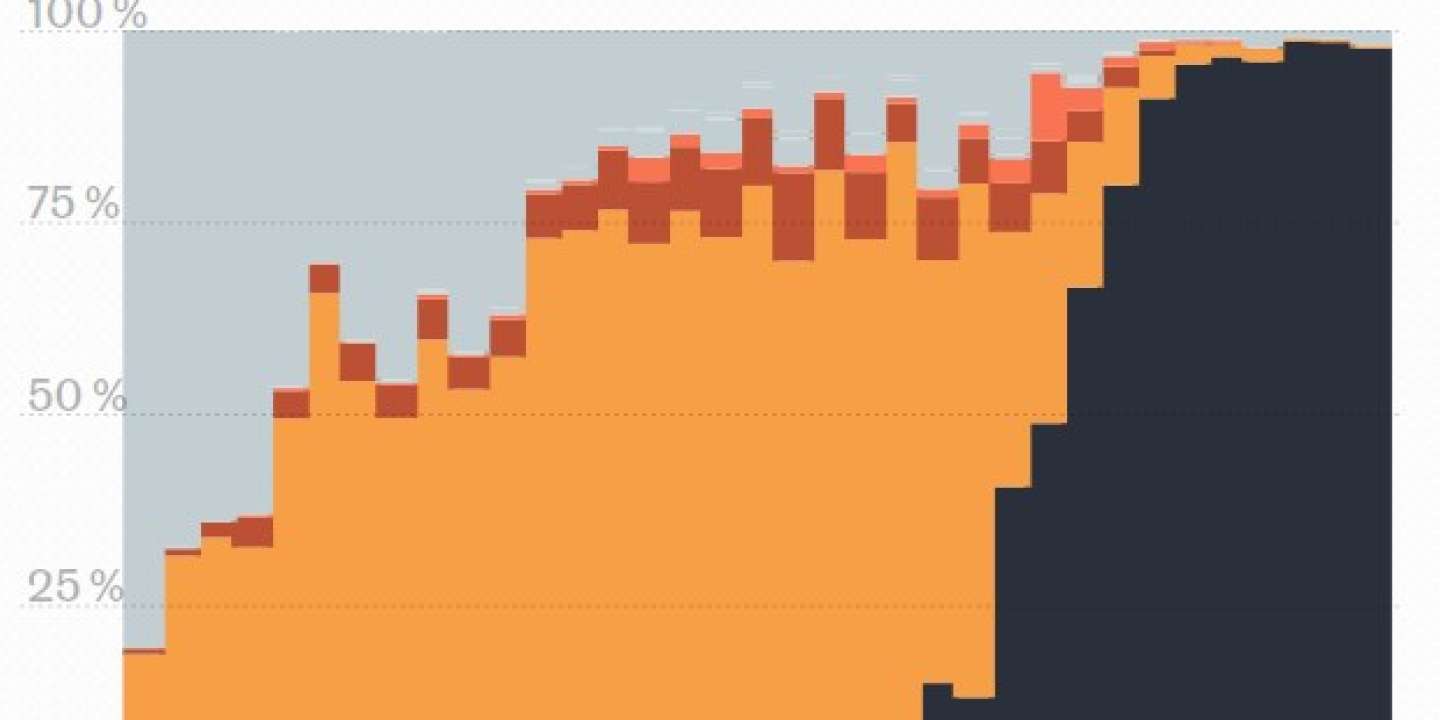
Il est possible d’observer leur circulation sur le territoire français grâce au séquençage : à partir de prélèvements RT-PCR de malades du Covid-19, cette technique de biologie moléculaire permet de lire le génome complet du SARS-CoV-2 et de détecter de nouvelles mutations. Les données peuvent ensuite être partagées sur la base internationale Global Initiative on Sharing Avian Influenza Data (Gisaid, pour « Initiative mondiale sur le partage des données relatives à la grippe aviaire »), dont le Royaume-Uni est l’un des premiers contributeurs.
La part estimée pour chaque variant reste approximative, car toutes les séquences ne sont pas déposées sur Gisaid, malgré les incitations du conseil scientifique. Il existe aussi d’autres biais (délai de publication, développement tardif du séquençage et évolutions de la stratégie nationale en la matière…), mais ces données, actualisées de façon hebdomadaire, montrent la quasi-hégémonie du variant Delta en France métropolitaine.
‘; else { color = “#C3CED2” } } else { color = “#C3CED2”; } return color }); document.getElementById(“d_legend_sequencage”).innerHTML += ‘
‘ + “Autre” + ‘
‘; let rect = groups.selectAll(“rect”) .data(d => d) .enter() .append(“rect”) .attr(“x”, function(d) { let date = getDateOfISOWeek(d.data.week.split(“-“)[1], d.data.week.split(“-“)[0]) return x(date)}) .attr(“width”, width_bar) .attr(“y”, d => y(d[1])) .attr(“height”, d => height – y(d[1] – d[0])); svg.append(“g”).attr(“class”, “axisBottom”).attr(“transform”, “translate(0,” + height + “)”).call( d3.axisBottom(x).tickSizeOuter(0).ticks(timeMonth > 6 ? d3.timeYear:d3.timeMonth, timeMonth > 6 ? 1:timeMonth) .tickFormat(function(n) { let exp = timeMonth == 12 ? date_axis(n, mobile,true, lastYear) : date_axis(n, mobile, false, lastYear); return exp })).selectAll(“text”).attr(‘class’, ‘d_axis_x_tickvalues’); svg_act.append(“g”).attr(“class”, “axisBottom”).attr(“transform”, “translate(0,” + height/5 + “)”).call( d3.axisBottom(x).tickSize(0).tickSizeOuter(0) // .ticks(timeMonth > 6 ? d3.timeYear:d3.timeMonth, timeMonth > 6 ? 1:timeMonth) .ticks(0) .tickFormat(function(n) { let exp = timeMonth == 12 ? date_axis(n, mobile,true, lastYear) : date_axis(n, mobile, false, lastYear); return exp })).selectAll(“text”).attr(‘class’, ‘d_axis_x_tickvalues’); svg_act.append(“text”) .attr(“x”, 0+margin.left) .attr(“y”, (height/5) + margin.bottom/2.5) .attr(‘class’, ‘d_axis_x_tickvalues’) .text(“Nombre de séquences disponibles”); svg.append(“g”).attr(“class”, “axisLeft”).call(d3.axisLeft(y) .tickValues(ticks) .tickSize(- ( width – margin.left – margin.right )) .tickFormat(function(n) { return numberWithSpace(n) + ” %”})).selectAll(“text”) .attr(‘class’, ‘d_axis_y_tickvalues’) .attr(“y”, -8).attr(“x”, 3).style(“text-anchor”, “start”); var max_cas = d3.max(sequencage_variants, function(d) { return d.number_sequenced }); var max_cas_round = Math.round(max_cas/1000 + 1) * 1000; var y_act = d3.scaleLinear().domain([0, max_cas_round]).range([ height/5, 0 ]); var ticks = [max_cas_round/4, max_cas_round/2, max_cas_round * 3/4, max_cas_round]; svg_act.append(“g”).attr(“class”, “axisLeft”).call(d3.axisLeft(y_act) .tickValues(ticks) .tickSize(- ( width – margin.left – margin.right )) .tickFormat(function(n) { return numberWithSpace(n)})).selectAll(“text”) .attr(‘class’, ‘d_axis_y_tickvalues’) .attr(“y”, -8).attr(“x”, 3).style(“text-anchor”, “start”); svg_act .selectAll(“.bar”) .data(sequencage_variants) .enter() .append(“rect”) .attr(“x”, function(d) { let date = getDateOfISOWeek(d.week.split(“-“)[1], d.week.split(“-“)[0]) return x(date); }) .attr(“y”, function(d) { return (y_act(d.number_sequenced)); }) .attr(“width”, (width – margin.left – margin.right) / (sequencage_variants.length )) .attr(“fill”,”#C3CED2″) .attr(“class”,”d_histo”) .attr(“height”, function(d) { return height/5 – y_act(d.number_sequenced); }) function numberWithSpace(x) { return x.toString().replace(/\B(?=(\d{3})+(?!\d))/g, ” “); } function date(n, day, short, year, only) { const options = { weekday: ‘long’, year: ‘numeric’, month: short == true ? “short” : ‘long’, day: ‘numeric’ }; var txt = n.toLocaleDateString(undefined, options).split(” “); let txt_year = year == false ? “” : ” ” + txt[3]; let txt_day = Number(txt[1]) == 1 ? “1er” : String(txt[1]); return only == true ? txt_year : day == false ? txt[2] + txt_year : txt_day + ” ” + txt[2] + txt_year ; } function date_axis(n, short, only, lastYear) { const options = { weekday: ‘long’, year: ‘numeric’, month: short == true ? “short” : ‘long’, day: ‘numeric’ }; var txt = n.toLocaleDateString(undefined, options).split(” “); let year_str = short == true ? txt[3].substring(2):txt[3]; if (only == true) return txt[3] else if (n.getMonth() + 1 == 1 && lastYear 2020) return txt[2].replace(“janv.”, “jan.”) + ” ” + year_str; else return txt[2] } function getDateOfISOWeek(w, y) { var simple = new Date(y, 0, 1 + (w – 1) * 7); var dow = simple.getDay(); var ISOweekStart = simple; if (dow
Le tableau ci-dessous recense les caractéristiques des principaux variants, selon la classification de l’OMS. Les plus « préoccupants » (ou VoC pour Variant of Concern) sont listés pour leur transmissibilité, leur virulence ou encore la moindre efficacité des vaccins face à eux. S’y ajoute une série de variants à suivre (VoI, pour Variant of Interest), qui pourraient avoir des conséquences sur la situation épidémiologique. Régulièrement, l’OMS ajoute de nouvelles souches ou déclasse certains variants, par exemple quand leur circulation diminue de façon substantielle.
Lignage
Circulation en FranceCirculation
Nom
Lignage
Circulation en FranceCirculation
` + d.name + `
` + lineage + `
` + d.circulation + `
` html_tab += “ return html_tab } var keys_variants = Object.keys(variants); let ct = 0 $.each(variants, function(i, d) { if (d.name != “” && d.type_oms == “VOC”) { mesure_clean = document.createElement(“div”); mesure_clean.innerHTML = d.detail; html_tab_VOC += generate_html_tab(i, d, keys_variants[ct]) } if (d.name != “” && d.type_oms == “VOI”) { mesure_clean = document.createElement(“div”); mesure_clean.innerHTML = d.detail; html_tab_VOI += generate_html_tab(i, d,keys_variants[ct]) } ct++ }); html_tab_VOC += “
” html_tab_VOI += “
” if (mobiledec) { var aulong = false } document.getElementById(“d_container_VOC”).innerHTML = html_tab_VOC document.getElementById(“d_container_VOI”).innerHTML = html_tab_VOI $(‘input#recherchemesure’).on(‘input’, function(e) { var mot = e.target.value.toLowerCase().normalize(“NFD”).replace(/[\u0300-\u036f]/g, “”) $(“.d_mesure”).show(); $(“.d_mesure.d_contenant”).hide(); $(“.d_mesure”).each(function(i, d) { var trouve = false; var id = d.dataset.id; for (const prop in d.dataset) { var corpus = d.dataset[prop].toLowerCase().normalize(“NFD”).replace(/[\u0300-\u036f]/g, “”) if (corpus.includes(mot)) { trouve = true; } } if (!trouve) { $(“.d_mesure[data-id=” + id + “]”).hide(); } else { $(“.d_ligne[data-id=” + id + “]”).show(); } }); }); /* on gère l’événement sur le chevron */ d3.selectAll(“#d_tab_variants .d_ligne:not(.entete)”).on(“click”, function() { toggleCase(this) }) function date(n, day, short, year, only) { const options = { weekday: ‘long’, year: ‘numeric’, month: short == true ? “short” : ‘long’, day: ‘numeric’ }; var txt = n.toLocaleDateString(undefined, options).split(” “); let txt_year = year == false ? “” : ” ” + txt[3]; let txt_day = Number(txt[1]) == 1 ? “1er” : String(txt[1]); return only == true ? txt_year : day == false ? txt[2] + txt_year : txt_day + ” ” + txt[2] + txt_year ; } function numberWithSpace(x) { return x.toString().replace(/\B(?=(\d{3})+(?!\d))/g, ” “);
} function toggleCase(ceci) { var classeChevronRestr = d3.select(ceci).select(“.chevron”).attr(“class”) /* on vérifie si c’est déjà déplie ou pas */ if (classeChevronRestr.indexOf(“bottom”) != -1) { d3.selectAll(“#d_tab_variants .d_contenant”).style(“display”, “none”) d3.selectAll(“#d_tab_variants .clic span.chevron”).classed(“bottom”, false) } else { var id = d3.select(ceci).attr(“data-id”); d3.selectAll(“#d_tab_variants .d_contenant”).style(“display”, “none”) d3.selectAll(“#d_tab_variants .clic span.chevron”).classed(“bottom”, false) d3.select(ceci).select(“span.chevron”).classed(“bottom”, true) d3.select(“#d_tab_variants .d_contenant[data-id='” + id + “‘]”).style(“display”, “block”) } } });
Plusieurs variants présentent des mutations communes jugées d’« intérêt » par les autorités sanitaires, « car elles semblent associées soit à une augmentation de la transmissibilité, soit à une diminution de l’efficacité de certains vaccins », expliquait, en juin, Sibylle Bernard-Stoecklin, de Santé publique France.
La technique de criblage, complémentaire au séquençage, permet de surveiller directement les mutations et leur évolution parmi les infections, au lieu de lire l’intégralité du génome du virus. Cette méthode ne permet pas d’identifier les variants, mais a l’avantage de pouvoir être employée à plus large échelle. Entre 40 % et 60 % des tests positifs au SARS-CoV-2 bénéficient ainsi d’un criblage chaque semaine. Les résultats montrent également la présence quasi exclusive du variant Delta, qui est l’un des porteurs de la mutation L452R.
‘ + ‘
‘ + mutations.B.name + “ (” + mutations.B.variants + ‘)
‘ + ‘
‘ + mutations.C.name + “ (” + mutations.C.variants + ‘)
‘; if (window.innerWidth = 2023) timeMonth = 12; else { let months = 12 * (lastYear – 2020) + lastMonth – 1; while (months/timeMonth > 6 || timeMonth % 3 != 0){ timeMonth += 1; } } } svg.append(“g”).attr(“class”, “axisBottom”).attr(“transform”, “translate(0,” + height + “)”).call( d3.axisBottom(x).tickSizeOuter(0).ticks(timeMonth > 6 ? d3.timeYear:d3.timeMonth, timeMonth > 6 ? 1:timeMonth) .tickFormat(function(n) { let exp = timeMonth == 12 ? date_axis(n, mobile,true, lastYear) : date_axis(n, mobile, false, lastYear); return exp })).selectAll(“text”).attr(‘class’, ‘d_axis_x_tickvalues’); svg.append(“g”).attr(“class”, “axisLeft”).call(d3.axisLeft(y) .tickValues(ticks) .tickSize(- ( width – margin.left – margin.right )) .tickFormat(function(n) { return numberWithSpace(n) + ” %”})).selectAll(“text”) .attr(‘class’, ‘d_axis_y_tickvalues’) .attr(“y”, -8).attr(“x”, 3).style(“text-anchor”, “start”); svg.append(“path”).datum(criblage_mutations) .attr(“fill”, “none”) .attr(“stroke”, mutations.A.color) .attr(“stroke-width”, 2) .attr(“d”, d3.line() .x(function(d) { return x(parseTime(d.week))}) .y(function(d) { return y(d.A1/(d.A1 + d.A0)*100)})); svg.append(“path”).datum(criblage_mutations) .attr(“fill”, “none”) .attr(“stroke”, mutations.B.color) .attr(“stroke-width”, 2) .attr(“d”, d3.line() .x(function(d) { return x(parseTime(d.week))}) .y(function(d) { return y(d.B1/(d.B1 + d.B0)*100)})); svg.append(“path”).datum(criblage_mutations) .attr(“fill”, “none”) .attr(“stroke”, mutations.C.color) .attr(“stroke-width”, 2) .attr(“d”, d3.line() .x(function(d) { return x(parseTime(d.week))}) .y(function(d) { return y(d.C1/(d.C1 + d.C0)*100)})); svg.selectAll(“.circle-points”).data(criblage_mutations) .enter() .append(“circle”) .attr(“stroke”, mutations.A.color) .attr(“fill”, “white”) .attr(“stroke-width”, “2px”) .attr(“id”, function(d, i) { return “d_A_” + String(i) }) .attr(“r”, function(d, i) { if(i == criblage_mutations.length – 1 || i == 0) return “3” }) .attr(“cx”,function(d) { return x(parseTime(d.week))}) .attr(“cy”, function(d) { return y(d.A1/(d.A1 + d.A0)*100)}); svg.selectAll(“.circle-points”).data(criblage_mutations) .enter() .append(“circle”) .attr(“stroke”, mutations.B.color) .attr(“fill”, “white”) .attr(“stroke-width”, “2px”) .attr(“id”, function(d, i) { return “d_B_” + String(i) }) .attr(“r”, function(d, i) { if(i == criblage_mutations.length – 1 || i == 0) return “3” }) .attr(“cx”,function(d) { return x(parseTime(d.week))}) .attr(“cy”, function(d) { return y(d.B1/(d.B1 + d.B0)*100)}) svg.selectAll(“.circle-points”).data(criblage_mutations) .enter() .append(“circle”) .attr(“stroke”, mutations.C.color) .attr(“fill”, “white”) .attr(“stroke-width”, “2px”) .attr(“id”, function(d, i) { return “d_C_” + String(i) }) .attr(“r”, function(d, i) { if(i == criblage_mutations.length – 1 || i == 0) return “3” }) .attr(“cx”,function(d) { return x(parseTime(d.week))}) .attr(“cy”, function(d) { return y(d.C1/(d.C1 + d.C0)*100)}) function numberWithSpace(x) { return x.toString().replace(/\B(?=(\d{3})+(?!\d))/g, ” “); } function date(n, day, short, year, only) { const options = { weekday: ‘long’, year: ‘numeric’, month: short == true ? “short” : ‘long’, day: ‘numeric’ }; var txt = n.toLocaleDateString(undefined, options).split(” “); let txt_year = year == false ? “” : ” ” + txt[3]; let txt_day = Number(txt[1]) == 1 ? “1er” : String(txt[1]); return only == true ? txt_year : day == false ? txt[2] + txt_year : txt_day + ” ” + txt[2] + txt_year ; } function date_axis(n, short, only, lastYear) { const options = { weekday: ‘long’, year: ‘numeric’, month: short == true ? “short” : ‘long’, day: ‘numeric’ }; var txt = n.toLocaleDateString(undefined, options).split(” “); let year_str = short == true ? txt[3].substring(2):txt[3]; if (only == true) return txt[3] else if (n.getMonth() + 1 == 1 && lastYear 2020) return txt[2].replace(“janv.”, “jan.”) + ” ” + year_str; else return txt[2] } })

